Vor etwa zwei Jahren habe ich Proteinstrukturen gelöst. Ich war mitten in der Bachelorarbeit und fleißig dabei Kristalle zu züchten, sie mit Röntgenstrahlen zu beschießen und aus dem Beugungsmuster die molekulare Struktur zu ermitteln.
Heute würde ich mich vielleicht lieber gleich an den PC setzen.
Denn das neue AlphaFold 3 Modell kann etwas, was AlphaFold 2 noch nicht konnte – DNA/RNA und kleine Moleküle, sogenannte Liganden, modellieren. Gerade an letzteren war ich damals interessiert. Ich wollte sehen, wie sie an mein Protein binden und an welcher Stelle.
Zum Spaß habe ich heute Morgen, während ich den Artikel schreibe, mein damaliges Protein in den neuen AlphaFold 3 Webserver geworfen. Das hat etwa eine Minute gedauert.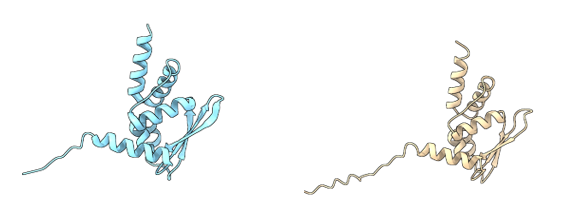
Links meine experimentelle Struktur, rechts die von AlphaFold 3 vorhergesagte Struktur.
Bei den kleinen Molekülen macht AlphaFold 3 uns aber erstmal einen Strich durch die Rechnung, denn Google DeepMind‘s Webserver bietet uns nur Zugang zu Klassikern wie z.B. ATP, NAD und Zitronensäure. AlphaFold 3 soll aber alle möglichen Liganden modellieren können! Und die Spinoff Firma Isomorphic Labs, gegründet von Google Deepmind CEO Demis Hassabis, wird wahrscheinlich gerade alle möglichen potenziellen Pharmaka modellieren und, wenn es gut läuft, ein Milliardengeschäft mit der in silico Vorhersage medizinischer Wirkstoffe machen.
Zunächst war das eine große Enttäuschung für mich. War doch AlphaFold 2 noch open source und verfügbar für jeden, schien Google jetzt die vollen Kapazitäten des Modells für die allgemeine wissenschaftliche Community aus ökonomischen Interessen zurückzuhalten. Das Supplement des Nature Papers sagt es selbst: „AlphaFold 3 wird für nicht-kommerzielle Zwecke als Server verfügbar. (…) Pseudocode zur Beschreibung der Algorithmen ist im Supplement verfügbar. Der Code ist nicht verfügbar.“
Das ist gewissermaßen ein Präzedenzfall für Nature. Denn bei einer Veröffentlichung muss auch der Code mitgeliefert werden. Daraufhin gab es einen offenen Brief an Nature, in dem kritisiert wurde, dass das Fehlen des open-source Codes unter anderem die Reproduzierbarkeit einschränkt. Immerhin hat DeepMind‘s Research Vizepräsident Pushmeet Kohli mittlerweile auf Social Media (X) gepostet, dass „an einer Veröffentlichung des AlphaFold 3 Modells (inklusive Weights) für akademische Zwecke“ gearbeitet werde. Weights sind hierbei die Konfigurationen, welche die KI bei ihrem Training erlernt.
Über wen soll ich mich jetzt eigentlich aufregen? Über Google, ohne deren gewaltiges Kapital und Know-How AlphaFold 3 wohlmöglich nie entstanden wäre? Oder über Nature, die zumindest versuchen, private Forschung für eine größere Allgemeinheit zu publizieren? Im schlimmsten Fall hätten wir keinen kostenlosen Zugang zur KI. Oder werden wir ihn verlieren, sobald wir von der Technologie abhängig sind?
Google legt die Ideen hinter den Algorithmen offen. Es ist nicht unwahrscheinlich, dass eine open-source Lösung entwickelt wird und Google lediglich ihrer Spinoff Firma Isomorphic Labs einen Vorsprung gibt.
Spannend ist doch, wie sich das Verhältnis zwischen Industrie und akademischen Sektor entwickelt. Viel Grundlagenforschung findet mittlerweile bei Firmen wie Google statt und die akademische Community bekommt einen Webservice für die spezifischeren Fragestellungen. Und was frage ich mich jetzt? Wenn ich die bestmögliche Forschung machen möchte, muss ich dann in den privaten Sektor?
Meinungen und Kommentare gerne an:
Quellen:
https://www.nature.com/articles/d41586-024-01463-0
https://x.com/pushmeet/status/1790086453520691657
https://www.nature.com/articles/s41586-024-07487-w